本文是上篇(特征选择 | 逻辑回归-通过系数符号和VIF进一步筛选变量)的续,是R版本的实现。
1、逐步选择,对R逻辑回归逐步选择变量的实现做了微调:
logit_stepwise <- function(data, label, slentry, slstay){
# author: 小石头(bigdata_0819@163.com)
# data:包含自变量和应变量的数据框
# label:应变量
# slentry:选择变量时显著水平判断的阈值
# slstay: 剔除变量时显著水平判断的阈值
# return: lst:
# SelectionProcess: 变量逐步选择的过程
# models: 每一步的模型结果
# model_optimal: 最终输出的模型
# result_optimal: 最终输出的模型结果
Selected <- c()
Remaining <- names(data)[which(names(data)!=label)]
NumberInModel <- 0
SelectionProcess_list <- list()
model_list <- list()
result_list <- list()
### 第0步,只有截距项
Step <- 0
formula <- paste0(label, ' ~ 1')
result <- glm(formula, data=data, family=binomial)
summ <- summary(result)
model <- data.frame(summ$coefficients)
model <- tibble::rownames_to_column(model, "Variable")
rownames(model) <- NULL
model <- cbind(Step, model)
colnames(model) <- c('Step', 'Variable', 'Estimate', 'StdErr', 'Zvalue', 'Pvalue')
model_list <- c(model_list, list(model))
### 逐步选择过程
while(length(Remaining)>0){
## 计算剩余变量的显著性
Remaining_Sl_list <- list()
for(var in Remaining){
formula <- paste0(label, ' ~ ', paste(c(Selected, var), collapse = "+"))
result <- glm(formula, data=data, family=binomial)
summ <- summary(result)
coef <- data.frame(summ$coefficients)
coef <- tibble::rownames_to_column(coef, "Variable")
coef <- coef[coef$Variable == var, ]
Remaining_Sl_list <- c(Remaining_Sl_list, list(coef))
}
Remaining_Sl <- dplyr::bind_rows(Remaining_Sl_list)
colnames(Remaining_Sl) <- c('Variable', 'Estimate', 'StdErr', 'Zvalue', 'Pvalue')
Entered_var <- Remaining_Sl$Variable[which.min(Remaining_Sl$Pvalue)]
z <- Remaining_Sl$Zvalue[which.min(Remaining_Sl$Pvalue)]
p <- min(Remaining_Sl$Pvalue)
if(p>slentry){
break
}else{
Step <- Step+1
NumberInModel <- NumberInModel+1
Selected <- c(Selected, Entered_var)
Remaining <- Remaining[Remaining!=Entered_var]
SelectionProcess <- data.frame(Step=Step,
Entered=Entered_var,
Removed='-',
NumberInModel=NumberInModel,
Zvalue=z,
Pvalue=p,
stringsAsFactors=FALSE)
SelectionProcess_list <- c(SelectionProcess_list, list(SelectionProcess))
formula <- paste0(label, ' ~ ', paste(Selected, collapse = "+"))
result <- glm(formula, data=data, family=binomial)
summ <- summary(result)
model <- data.frame(summ$coefficients)
model <- tibble::rownames_to_column(model, "variable")
rownames(model) <- NULL
model <- cbind(Step, model)
colnames(model) <- c('Step', 'Variable', 'Estimate', 'StdErr', 'Zvalue', 'Pvalue')
model_list <- c(model_list, list(model))
result_list <- c(result_list, list(result))
## 剔除已选择变量中最不显著的变量
var_Pvalue <- model[model$Variable!='(Intercept)', ]
if(max(var_Pvalue$Pvalue)>slstay){
Step <- Step+1
NumberInModel <- NumberInModel-1
Removed_var <- var_Pvalue$Variable[which.max(var_Pvalue$Pvalue)]
z <- var_Pvalue$Zvalue[which.max(var_Pvalue$Pvalue)]
p <- max(var_Pvalue$Pvalue)
Selected <- Selected[Selected!=Removed_var]
Remaining <- c(Remaining, Removed_var)
SelectionProcess <- data.frame(Step=Step,
Entered='-',
Removed=Removed_var,
NumberInModel=NumberInModel,
Zvalue=z,
Pvalue=p,
stringsAsFactors=FALSE)
SelectionProcess_list <- c(SelectionProcess_list, list(SelectionProcess))
formula <- paste0(label, ' ~ ', paste(Selected, collapse = "+"))
result <- glm(formula, data=data, family=binomial)
summ <- summary(result)
model <- data.frame(summ$coefficients)
model <- tibble::rownames_to_column(model, "variable")
rownames(model) <- NULL
model <- cbind(Step, model)
colnames(model) <- c('Step', 'Variable', 'Estimate', 'StdErr', 'Zvalue', 'Pvalue')
model_list <- c(model_list, list(model))
result_list <- c(result_list, list(result))
}
}
}
SelectionProcess <- dplyr::bind_rows(SelectionProcess_list)
models <- dplyr::bind_rows(model_list)
model_optimal <- model_list[[length(model_list)]]
result_optimal <- result_list[[length(result_list)]]
model_optimal$significance[model_optimal$Pvalue<=0.001] <- '***'
model_optimal$significance[model_optimal$Pvalue>0.001 & model_optimal$Pvalue<=0.01] <- '**'
model_optimal$significance[model_optimal$Pvalue>0.01 & model_optimal$Pvalue<=0.05] <- '*'
model_optimal$significance[model_optimal$Pvalue>0.05] <- ''
lst <- list(SelectionProcess=SelectionProcess, models=models, model_optimal=model_optimal, result_optimal=result_optimal)
return(lst)
}
2、迭代剔除系数为负值和VIF较大的变量:
model_logit <- function(data, label, slentry, slstay){
# author: 小石头(bigdata_0819@163.com)
# data:包含自变量和应变量的数据框
# label:应变量
# return: lst:
# SelectionProcess: 变量逐步选择的过程
# models: 每一步的模型结果
# model_optimal: 最终输出的模型
# result_optimal: 最终输出的模型结果
while(TRUE){
### 剔除系数为负值的变量
while(TRUE){
lst <- logit_stepwise(data=data, label=label, slentry=slentry, slstay=slstay)
SelectionProcess <- lst$SelectionProcess
models <- lst$models
model_optimal <- lst$model_optimal
result_optimal <- lst$result_optimal
vars_minus <- model_optimal$Variable[model_optimal$Estimate<0]
vars_minus <- SelectionProcess$Entered[SelectionProcess$Entered %in% vars_minus]
if(length(vars_minus)>0){
data <- dplyr::select(data, -c(vars_minus[length(vars_minus)]))
}else{
break
}
}
### 剔除VIF>10的变量
model_vars <- model_optimal$Variable[model_optimal$Variable!='(Intercept)']
formula = paste0(label, ' ~ ', paste(model_vars, collapse = "+"))
la <- lm(formula, data)
VIF <- car::vif(la)
if(max(VIF)>10){
data <- dplyr::select(data, -c(names(VIF)[which.max(VIF)]))
}else{
break
}
}
VIF_df <- data.frame(Variable=names(VIF), VIF=VIF)
model_optimal <- merge(model_optimal, VIF_df, by='Variable', all.x=TRUE, all.y=FALSE)
model_optimal <- dplyr::select(model_optimal, -c('Step'))
Step_df <- SelectionProcess[!duplicated(SelectionProcess$Entered, fromLast=TRUE), c('Step', 'Entered')]
colnames(Step_df) <- c('Step', 'Variable')
model_optimal <- merge(Step_df, model_optimal, by='Variable', all.x=FALSE, all.y=TRUE)
model_optimal[model_optimal$Variable=='(Intercept)', 'Step'] <- 0
model_optimal <- dplyr::arrange(model_optimal, Step)
model_optimal$Step <- 1:length(model_optimal$Step)-1
lst <- list(SelectionProcess=SelectionProcess, models=models, model_optimal=model_optimal, result_optimal=result_optimal)
return(lst)
}
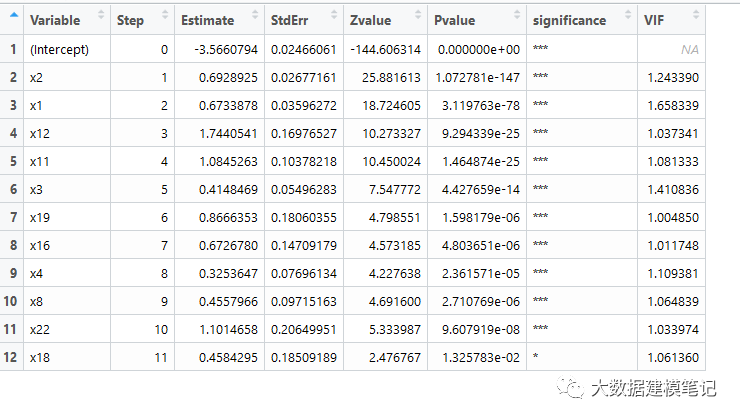
使用相同数据集查看训练结果,可以看出最终结果和上篇的一致的。
lst <- model_logit(data=data, label='target', slentry=0.05, slstay=0.05)
model_optimal <- lst$model_optimal
![]()
同时发现VIF的值有一些差异,主要因为这里使用的R方法在计算VIF做线性回归时包含了截距项,而Python模块statsmodels.stats.outliers_influence中的variance_inflation_factor函数在做线性回归时没有包含截距项(见下面的源码)。是否包含截距项对VIF值的影响不大,也不改变各变量VIF的排序性,因而并不影响结果。
variance_inflation_factor的源码:
##### variance_inflation_factor源码 #####
"""Influence and Outlier Measures
Created on Sun Jan 29 11:16:09 2012
Author: Josef Perktold
License: BSD-3
"""
from statsmodels.regression.linear_model import OLS
def variance_inflation_factor(exog, exog_idx):
"""variance inflation factor, VIF, for one exogenous variable
The variance inflation factor is a measure for the increase of the
variance of the parameter estimates if an additional variable, given by
exog_idx is added to the linear regression. It is a measure for
multicollinearity of the design matrix, exog.
One recommendation is that if VIF is greater than 5, then the explanatory
variable given by exog_idx is highly collinear with the other explanatory
variables, and the parameter estimates will have large standard errors
because of this.
Parameters
----------
exog : ndarray
design matrix with all explanatory variables, as for example used in
regression
exog_idx : int
index of the exogenous variable in the columns of exog
Returns
-------
vif : float
variance inflation factor
Notes
-----
This function does not save the auxiliary regression.
See Also
--------
xxx : class for regression diagnostics TODO: does not exist yet
References
----------
https://en.wikipedia.org/wiki/Variance_inflation_factor
"""
k_vars = exog.shape[1]
x_i = exog[:, exog_idx]
mask = np.arange(k_vars) != exog_idx
x_noti = exog[:, mask]
r_squared_i = OLS(x_i, x_noti).fit().rsquared
vif = 1. / (1. - r_squared_i)
return vif
特征选择 | 逻辑回归-通过系数符号和VIF进一步筛选变量
WOE(证据权重)为何这样计算?
Vintage损失率转为年化损失率(名义利率/内部收益率/年化利率等概念)